best365网页版登录蒋超实验室在Briefings in Bioinformatics杂志发表了题为“iPhylo: An Automated and Interactive Platform for Biological and Chemical Taxonomic Analysis”的研究论文。该工作开发了名为iPhylo的一个集成生物与化学分类分析、可视化和注释的自动化平台(https://www.iphylo.net/)。
准确快速的分类学分析对于系统探索多样环境中的生物和代谢产物至关重要。现有的生物学和化学分类工具大多只能处理单一领域的数据,缺乏有效的整合分析平台,且在用户交互性、可定制性和跨平台操作方面存在局限性。快速准确的系统发育树和代谢相关化合物分类树的构建对微生物组学,代谢组学,多组学,生态学和环境科学等研究领域具有非常重要的研究意义。
研究团队开发的iPhylo平台提供了一个统一的、跨学科的框架,支持生物学与化学的分类学分析。iPhylo不仅能处理来自NCBI的生物学数据,还能够结合ChemOnt、NPClassifier等化学分类系统,对化合物分类进行层级分析,并通过交互式的可视化平台简化了分类树注释过程,优化了可视化工作的共享与协作。
研究团队在不同规模的测试数据集上对iPhylo工具集与其他流行的系统发育分析工具和化合物分类工具(phyloT,iTOL,ChemTreeMap, Qemistree等)进行了性能测试与功能比对。iPhylo显示出其独特的功能性,定制性和灵活性,尤其是独特的生物和化学数据的整合分析能力。论文中展示了iPhylo平台在以下4个案例中的应用:1.冰川微生物基因组分析;2.人类肠道微生物的代谢谱分析;3.生物环境暴露组的分析;4.自定义数据集处理。总体而言,iPhylo工具集以其统一且交互的框架,在生物和化学特征的综合分析方面展现出显著优势,特别适用于微生物组学、环境科学以及精准医学等领域的多组学数据分析。
博士生李悦耳,彭晨为论文的共同第一作者,蒋超研究员为论文的通讯作者。参与该研究的成员还包括浙江大学长三角智慧绿洲创新中心研究员迟飞,蒋超实验室的博士生黄子诺,袁梦一,斯坦福大学研究员周欣. 该研究得到了国家自然科学基金等项目资助,以及生命科学研究院NECHO高性能计算集群的支持。
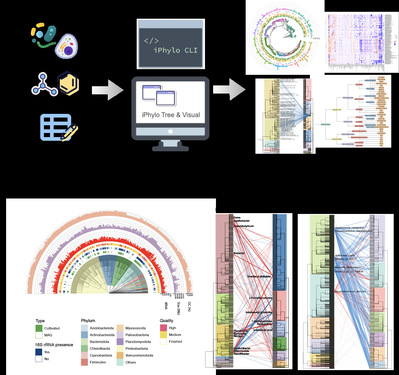
图1 iPhylo 工作流程